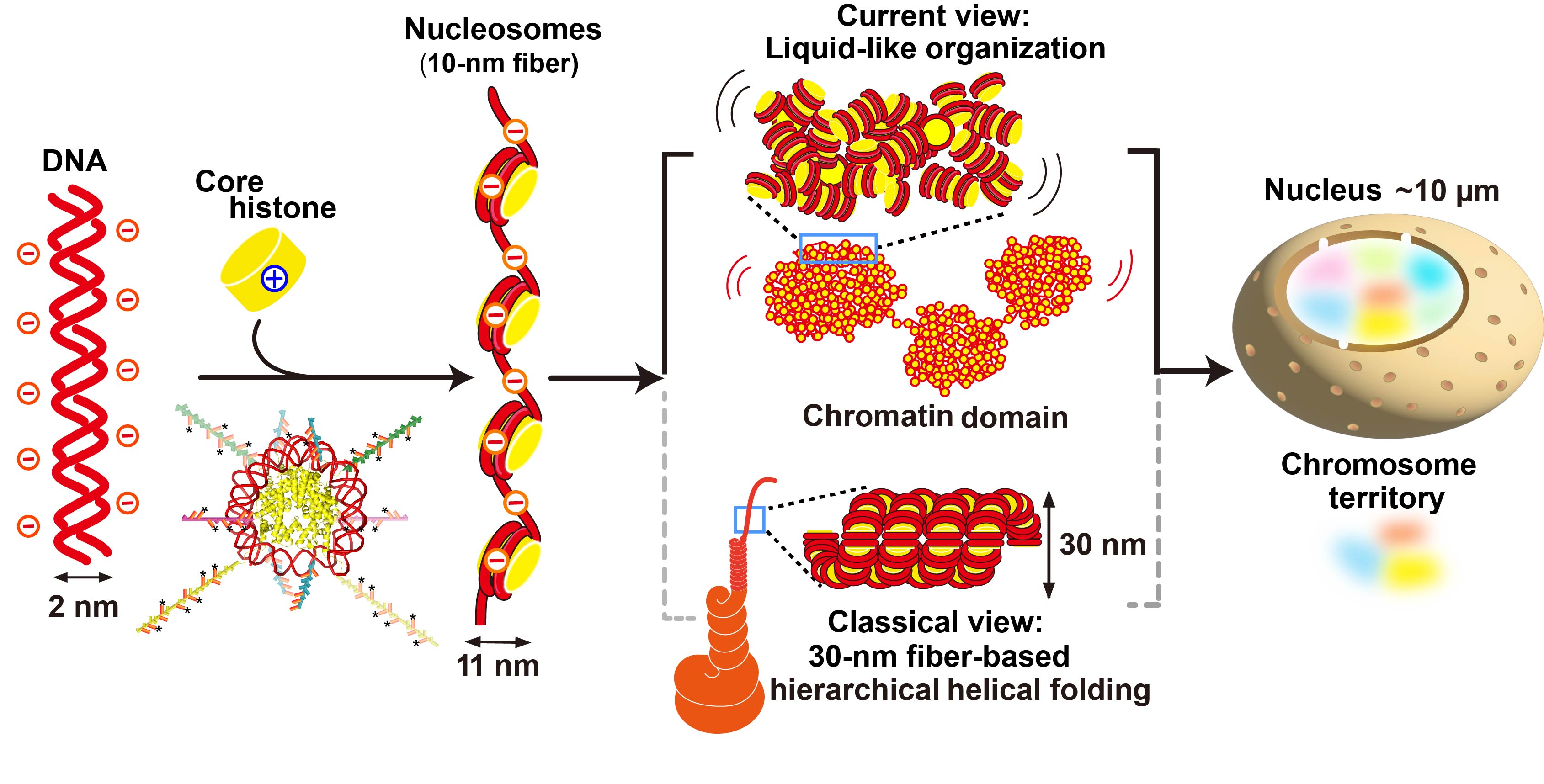
私たちの研究の背景
従来の説では、ヌクレオソームは規則正しく配置されて直径30 nmの線維をつくり、さらにらせん状に折り畳まれて階層構造を形成するとされていました
(図1下段)。この説に従うと、あるタンパク質がゲノムDNAのある領域に結合したい場合、すべての階層構造をほどき直す必要があります。こんなことが本当に細胞のなかで起こっているのでしょうか?
私たちは20年以上前からこの問題に取り組んできました。そして、ヒト細胞内のヌクレオソームは、30 nm線維をはじめとする規則正しい階層構造をつくらず、むしろ不規則に折り畳まれていることを明らかにしました
(図1上段)(Maeshima et al., Curr Opin Cell Biol2019)。
この不規則な折り畳みから、私たちはヌクレオソームが物理的にあまり束縛されておらず、流動的であると予想しました。実際、単一の分子を観測できる一分子顕微鏡を用いて、生きた細胞内の個々のヌクレオソームの動きを追跡すると、ヌクレオソームはその場でダイナミックに揺らぎ
(図2左、動画)、短い時間においては液体のように振る舞うことが判明しています (Iida et al., Science Adv 2022; Nozaki et al., Science Adv 2023)。また、超解像顕微鏡により、細胞内のヌクレオソームの分布を高解像度で観察することで、これらが直径200 nm程度のクロマチンドメインという塊を形成することも示してきました(図2右)(Nozaki
et al., Mol Cell 2017; Nozaki et al., Science Adv2023; Iida, Shimazoe et al. bioRxiv2025)。
DNAの複製やRNAへの転写などは、クロマチンドメインを単位として進行すると考えられています。さらに、これらのプロセスに関わるタンパク質が、クロマチンドメイン内部のDNA領域を探索するとき、ヌクレオソームの揺らぎがクロマチンドメイン内部へのアクセスを助けていることも明らかにしてきました
(Hihara et al., Cell Rep 2012)。

図2 左上の一個一個のドットは単一のヌクレオソームをあらわしている。各ドットの中心を正確に決定することにより、ヌクレオソームの動きを10 nm以下の精度で測定できる (Minami et al., Science Adv 2025)。右は、構造化照明顕微鏡による超解像クロマチン像。ここではユークロマチンが塊 (ドメイン) を形成している (Iida, Shimazoe et al., bioRxiv 2025)。
1分子ヌクレオソーム動画 (50 ms/frame) 一個一個のドットは単一のヌクレオソームをあらわし、ヌクレオソームが揺らいでいる。 Iida et al., Science Adv 2022から再掲。
研究課題
現在、私たちは生きた細胞において、クロマチンドメインの実態、すなわち、その物理的な性質を解明すること、さらに、クロマチンドメインがDNAの複製、修復、RNAへの転写などのゲノムの機能にどのように寄与するかを明らかにしようとしています。進行中のプロジェクトは以下の通りです。
新しい超解像顕微鏡システムの構築
生きた細胞のクロマチンドメインを可視化できる構造化照明顕微鏡と、一分子の動きを追跡できる顕微鏡を組み合わせ、新しい超解像顕微鏡システムを構築しています (図2)。このシステムを用いて、生きたヒト細胞のクロマチンドメインの構造と、その内部や外部のヌクレオソーム、タンパク質分子の振る舞いを観察しています(図3)(Shimazoe et al., bioRxiv2025)。
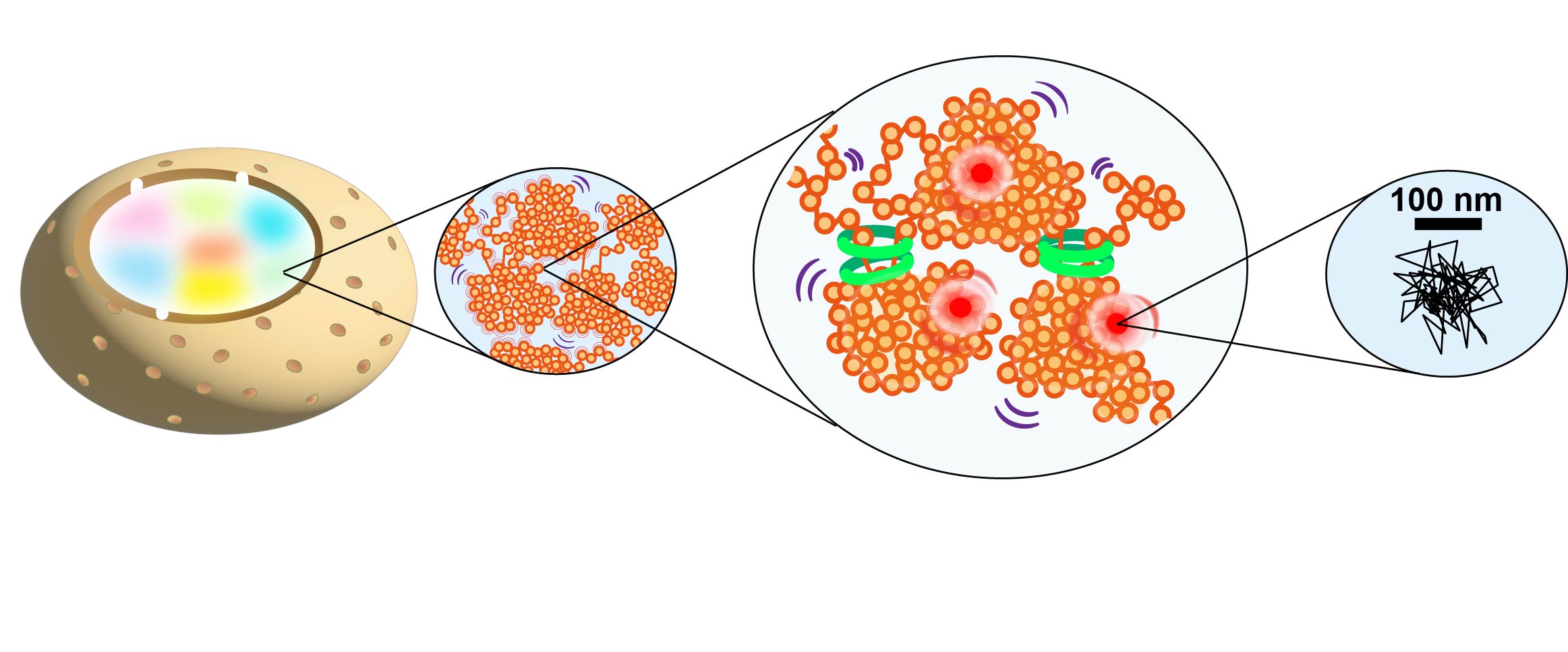
図3 生きた細胞のユークロマチン・ヘテロクロマチンのドメインの構造と、その中のヌクレオソームや他のタンパク質の動きを新規顕微鏡システムで観察する。そしてヌクレソームやタンパク質の軌跡を解析する(右)。
ユークロマチン・ヘテロクロマチンの特異的な標識方法の開発
クロクロマチンドメインの物理的な性質という側面から、転写制御などの細胞機能の理解、さらに、正常細胞、種々のがん細胞 (Otsuka et al.,
Cell Struct Function 2026)、老化細胞、疾患細胞を用い、クロマチンドメインの物理的な性質の変化を介した細胞機能不全・細胞分化のメカニズム解明を目指しています。マチンには遺伝子が活発に転写されているユークロマチンと、遺伝子が少なく、ほとんど不活化されているヘテロクロマチンが存在します。両者のヒストン修飾の違いなど、生化学的な相違はよく分かっている一方で、クロマチンドメインの物理的な性質の違いはほとんど分かっていません。このため、生きた細胞で両者を特異的に蛍光標識する方法Repli-Histo標識を開発しました
(図4) (Minami et al.,Science Adv 2025)。
さらに、生きた細胞のユークロマチン・ヘテロクロマチンドメインと、クロマチンドメインの内部や外部のヌクレオソーム、タンパク質分子の動きの軌跡を、構造化照明と一分子顕微鏡
(図2)を用いて測定しようとしています(図3)。各クロマチンドメインの物理的な性質と、クロマチンドメイン内部にタンパク質がどのようにアクセスするかを解析しています。さらに、実験と計算機モデルを組み合わせ、クロマチンドメインの実態とその形成メカニズムの解明をおこなっています。
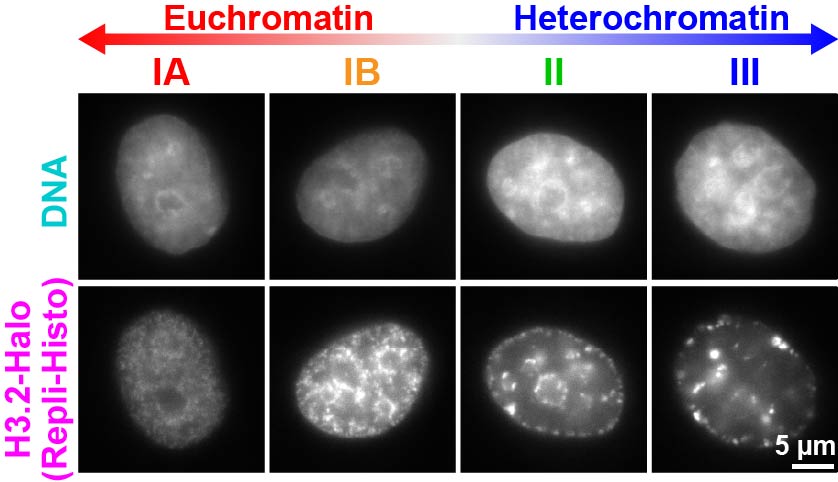
図4 ユークロマチンとヘテロクロマチンを標識できるRepli-Histo標識
上段はDAPIによるDNA染色、下段はRepl-Histo標識。クロマチンをクラスIA, IB (ユークロマチン)、クラスII, III (ヘテロクロマチン)と4段階に標識できる(Minami
et al. Science Adv2025)。
クロマチンドメインによる細胞機能の制御の理解
クロマチンドメインの物理的な性質という側面から、転写制御などの細胞機能の理解、さらに、正常細胞、種々のがん細胞 (Otsuka et al., Cell Struct Function 2026)、老化細胞、疾患細胞を用い、クロマチンドメインの物理的な性質の変化を介した細胞機能不全・細胞分化のメカニズム解明を目指しています。
References
1. Maeshima K. (2025) The shifting paradigm of chromatin structure: from
the 30-nm chromatin fiber to liquid-like organization. Proc Jpn Acad Ser B Phys Biol Sci 101:339–356. doi:10.2183/pjab.101.020.
2. Maeshima K, Ide S, Babokhov M. (2019) Dynamic chromatin organization
without the 30-nm fiber. Curr Opin Cell Biol58:95–104. doi:10.1016/j.ceb.2019.02.003.
3. Iida S, Shinkai S, Itoh Y, Tamura S, Kanemaki MT, Onami S, Maeshima
K. (2022) Single-nucleosome imaging reveals steady-state motion of interphase
chromatin in living human cells. Science Adv8: eabn5626. doi:10.1126/sciadv.abn5626.
4. Nozaki T, Imai R, Tanbo M, Nagashima R, Tamura S, Tani T, Joti Y, Tomita
M, Hibino K, Kanemaki MT, Wendt KS, Okada Y, Nagai T, Maeshima K. (2017)
Dynamic organization of chromatin domains revealed by super-resolution
live-cell imaging. Mol Cell 67: 282–293.e7. doi:10.1016/j.molcel.2017.06.018.
5. Nozaki T, Shinkai S, Ide S, Higashi K, Tamura S, Shimazoe MA, Nakagawa
M, Suzuki Y, Okada Y, Sasai M, Onami S, Kurokawa K, Iida S, Maeshima K.
(2023) Condensed but liquid-like domain organization of active chromatin
regions in living human cells. Science Adv 9: eadf1488. doi:10.1126/sciadv.adf1488.
6. Iida, S., Shimazoe, M.A., Minami, K., Tamura, S., Ashwin, S.S., Higashi,
K., Nishiyama, T., Kanemaki, M.T., Sasai, M., Schermelleh, L., Toyoda,
A., Kurokawa, K., Maeshima, K. (2025) Cohesin prevents local mixing of
condensed euchromatic domains in living human cells. bioRxiv. doi:10.1101/2025.08.27.672592.
7. Hihara S, Pack CG, Kaizu K, Tani T, Hanafusa T, Nozaki T, Takemoto S,
Yoshimi T, Yokota H, Imamoto N, Sako Y, Kinjo M, Takahashi K, Nagai T,
Maeshima K. (2012) Local nucleosome dynamics facilitate chromatin accessibility
in living mammalian cells. Cell Rep 2:1645–1656. doi:10.1016/j.celrep.2012.11.008.
8. Minami K, Nakazato K, Ide S, Kaizu K, Higashi K, Tamura S, Toyoda A,
Takahashi K, Kurokawa K, Maeshima K. (2025) Replication-dependent histone
labeling dissects the physical properties of euchromatin/heterochromatin
in living human cells. Science Adv 11: eadu8400. doi:10.1126/sciadv.adu8400.
9. Shimazoe MA, Huertas J, Phillips C, Ide S, Tamura S, Farr S, Ashwin
SS, Sasai M, Collepardo-Guevara R, Maeshima K. (2025) Linker histone H1
functions as a liquid-like glue to organize chromatin in living human cells.
bioRxiv. doi:10.1101/2025.03.05.641622.
10. Otsuka A, Shimazo MA, Watanabe S, Minami K, Tamura S, Kiyono T, Takeshita
F, Maeshima K. (2026) Cell Structure and Function. doi:10.1247/csf.25147